df <- read.csv(file = "Bases/sal_tension_arterial.csv")Regresión lineal simple
Introducción
Muchos de los aspectos teóricos serán explicados en la clase utilizando la siguiente presentación. Aquí nos centramos en como realizar una regresión lineal en R.
Existe e evidencia suficiente para conocer que el consumo de sal tiene efectos sobre la tensión arterial. A mayor consumo de sal se observan mayores niveles de tensión arterial. Un análisis de regresión lineal, entre otras cosas, permite cuantificar la relación entre dos variables. En este caso, se pretende cuantificar la relación entre el consumo de sal y la tensión arterial. Es decir, mediante un análisis de regresión lineal se puede establecer una relación matemática entre el consumo de sal y la tensión arterial y describir como cambian un variable en función de la otra.
Las técnicas de regresión se utilizan para:
- Predicción
- Identificación de factores de riesgos
- Para aislar el efecto de una sola variable
- Para identificar el efecto de varias variables sobre una dependiente
- Identificar la independencia de dos variables
- Análisis de supervivencia
Las técnicas de regresión son una de las técnicas más maravillosas que existen. Por lo pronto nos enfocaremos en su versión más simple: la regresión lineal simple.
Regresión lineal simple
El análisis de regresión lineal simple es una técnica que se utiliza para explicar o modelar la relación entre dos variables numéricas. En esta técnica se debe diferenciar e identificar la variable dependiente (y) y la variable independiente o predictora (x). Vamos a poner en contexto a la regresión lineal utilizando el ejemplo de la relación entre el consumo de sal y la tensión arterial.
La base de datos sal.csv contiene información sobre el consumo de sal y la tensión arterial. La variable sal representa el consumo de sal en mg y la variable tension representa la tensión arterial en mmHg.
Su objetivo es conocer la relación de estas dos variables. Para ello, se ajustará un modelo de regresión lineal simple.
Importación de los datos
Visaluación de los datos
Un punto importante antes de realizar una regresión es observar la relación que tienen los datos visualmente y medienta un coeficiente de correlación.
library(ggplot2)
df|>
ggplot(mapping = aes(x=Consumo_Sal, y=Tension_Arterial))+
geom_point(col= "cyan4", alpha = 0.8, size = 3)+
theme_linedraw()+labs(x="Comsumo de sal", y= "mmHg")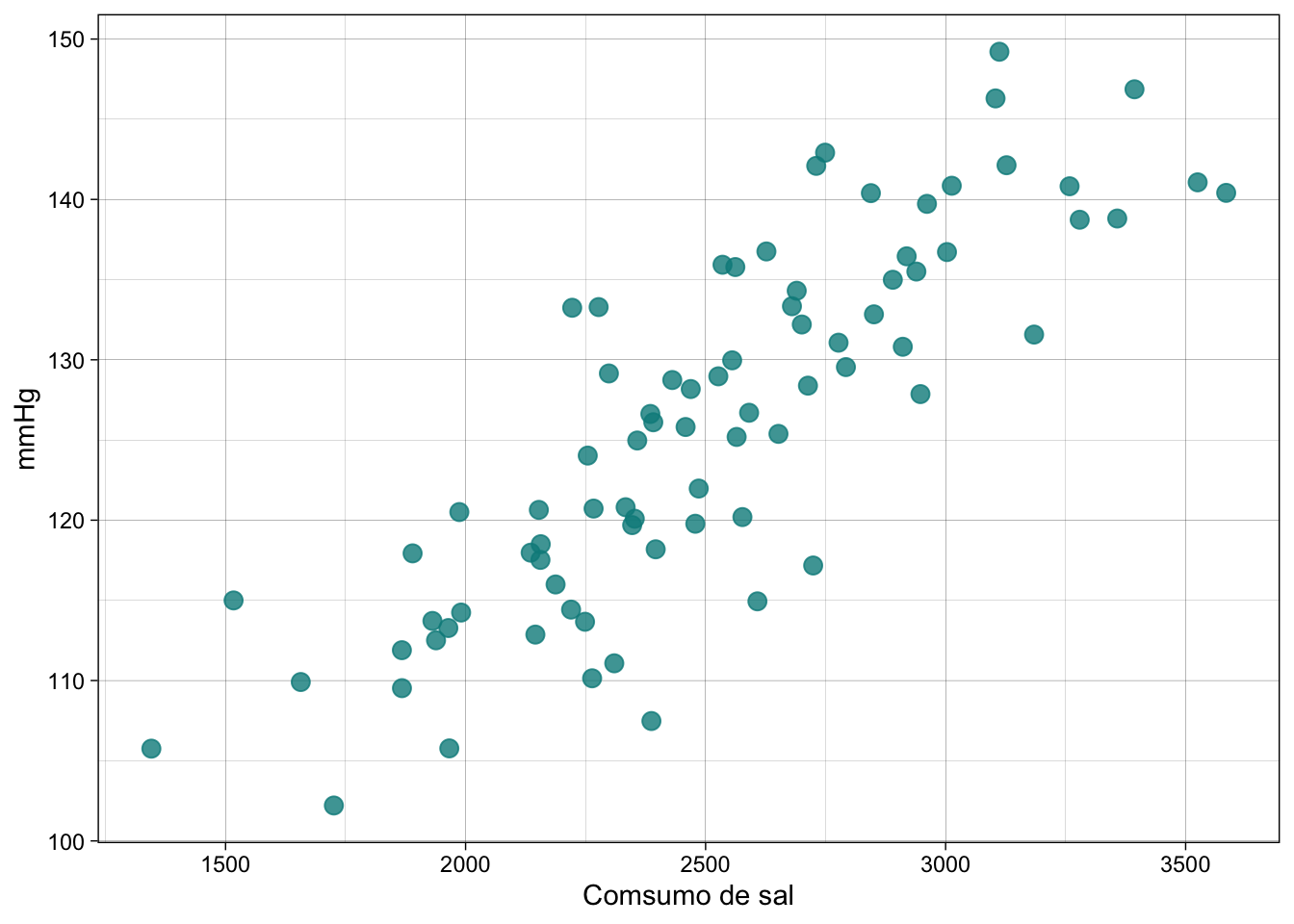
cor.test(df$Consumo_Sal, df$Tension_Arterial)
Pearson's product-moment correlation
data: df$Consumo_Sal and df$Tension_Arterial
t = 13.459, df = 78, p-value < 2.2e-16
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
0.7550534 0.8919377
sample estimates:
cor
0.8360682 Creación del modelo de regresión
lm(Tension_Arterial~Consumo_Sal, data = df)|>
summary()
Call:
lm(formula = Tension_Arterial ~ Consumo_Sal, data = df)
Residuals:
Min 1Q Median 3Q Max
-15.989 -3.424 -0.313 3.531 13.142
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 74.839105 3.861251 19.38 <2e-16 ***
Consumo_Sal 0.020370 0.001513 13.46 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 6.213 on 78 degrees of freedom
Multiple R-squared: 0.699, Adjusted R-squared: 0.6952
F-statistic: 181.1 on 1 and 78 DF, p-value: < 2.2e-16Lo primero que observamos en estos resultados es la ecuación de la recta: lm(formula = Tension_Arterial ~ Consumo_Sal, data = df). La interpretación de estos resultados requiere de la revisión de otros conceptos y de una descripción más detalla. De momento identifique el valor de 0.020370, el cual nos dice que por cada mg que se aumente el consumo de sal en una unidad, la tensión arterial aumenta en en promedio 0.020370 mmHg.
Interpretación de los resultados
Coeficientes \(\beta\)
En una regresión lineal simple hay dos coeficientes que son muy útiles para la interpretación correcta de los resultados. Estos dos valores son coeficientes \(\beta\) que son representados como \(\beta_1\) y \(\beta_0\) respectivamente.
Pendiente \(\beta_1\)
El coeficiente \(\beta_1\) es el valor que nos dice cuánto aumenta la variable dependiente por cada unidad que aumenta la variable independiente. En este caso, el coeficiente \(\beta_1\) es de 0.020370. Esto significa que por cada mg que se aumente el consumo de sal, la tensión arterial aumenta en promedio 0.020370 mmHg.
Intercepto
El intercepto \(\beta_0\) es valor en el que la linea de la regresión cruza el eje de las y. En otras palabras sería el valor de la presión arterial cuando el consumo de sal es cero, en este caso el intercepto es de 74.839105 mmHg. Tome en cuenta que esto no siempre tiene sentido.
Intervalos de confianza
lm(Tension_Arterial~Consumo_Sal, data = df)|>
confint() 2.5 % 97.5 %
(Intercept) 67.15194384 82.52626615
Consumo_Sal 0.01735684 0.02338306Para \(\beta_1\) los valores van de 0.017 a 0.023, no incluyen al 0. Por lo tanto podríamos asegurar que nuestro valor de \(\beta_1\) es diferente de 0 con un 95% de confianza.
Coeficiente de determinación
Pata este modelo \(R^2\) fue 0.699 (se puede obtener mediante summary), lo que significa que aproximadamente el 69.9% de la variabilidad en la tensión arterial puede ser explicada por el consumo de sal. El \(R^2\) puede tomar valores de 0 a 1
ANOVA
El ANOVA determina si existe una relación lineal significativa entre las variables independientes y la variable dependiente. La \(H_0\) para este ANOVA es que todos los coeficientes de regresión (excepto el intercepto) son iguales a cero
Se obtiene mediante summary
Evaluación de los supuestos
La regresión requiere para ser válida ciertos supuestos.
Normalidad de los residuos
En una regresión lineal, aunque sería ideal, no es necesario que los datos sigan una distribución normal. Lo que se requiere es que los residuos sigan una distribución normal. Esta normalidad se puede evaluar gráficamente, en el propio modelo de regresión y mediante pruebas estadísticas.
lm(Tension_Arterial~Consumo_Sal, data = df)|>
summary()
Call:
lm(formula = Tension_Arterial ~ Consumo_Sal, data = df)
Residuals:
Min 1Q Median 3Q Max
-15.989 -3.424 -0.313 3.531 13.142
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 74.839105 3.861251 19.38 <2e-16 ***
Consumo_Sal 0.020370 0.001513 13.46 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 6.213 on 78 degrees of freedom
Multiple R-squared: 0.699, Adjusted R-squared: 0.6952
F-statistic: 181.1 on 1 and 78 DF, p-value: < 2.2e-16modelo <- lm(Tension_Arterial~Consumo_Sal, data = df)
nortest::lillie.test(modelo$residuals)
Lilliefors (Kolmogorov-Smirnov) normality test
data: modelo$residuals
D = 0.061958, p-value = 0.6297library(ggrain)Registered S3 methods overwritten by 'ggpp':
method from
heightDetails.titleGrob ggplot2
widthDetails.titleGrob ggplot2ggplot(mapping = aes(x=1, y=modelo$residuals))+
geom_rain(fill="cyan4", alpha=0.5, col="black" )+
theme_light()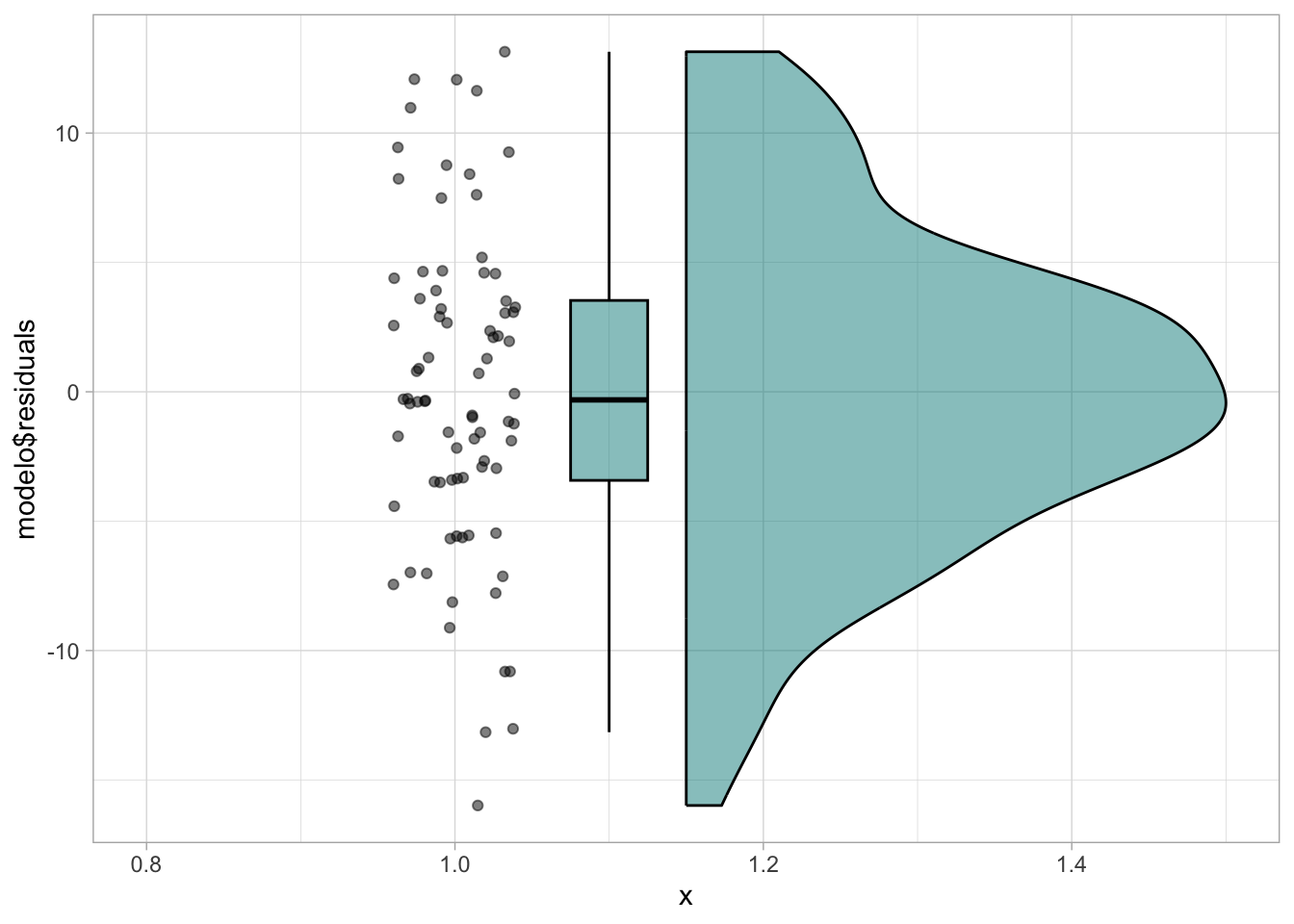
Evaluación gráfica de los modelos
Necesitamos evaluar gráficamente los supuestos de la regresión lineal. Para ello, podemos utilizar los siguientes gráficos:
- Gráfico de Residuos vs. Valores Ajustados: Permite evaluar la homoscedasticidad de las varianzas
- Gráfico Q-Q de Residuos: Permite evaluar la normalidad de los residuos
- Gráfico de Escala-Localización (Spread-Location Plot): Para evluar homoscedasticidad de las varianzas
- Residuos vs Leverage: Indentificación de valore atípicos
Estos gráficos se pueden obtener mediante la función plot del modelo de regresión y luego dar enter.
plot(modelo)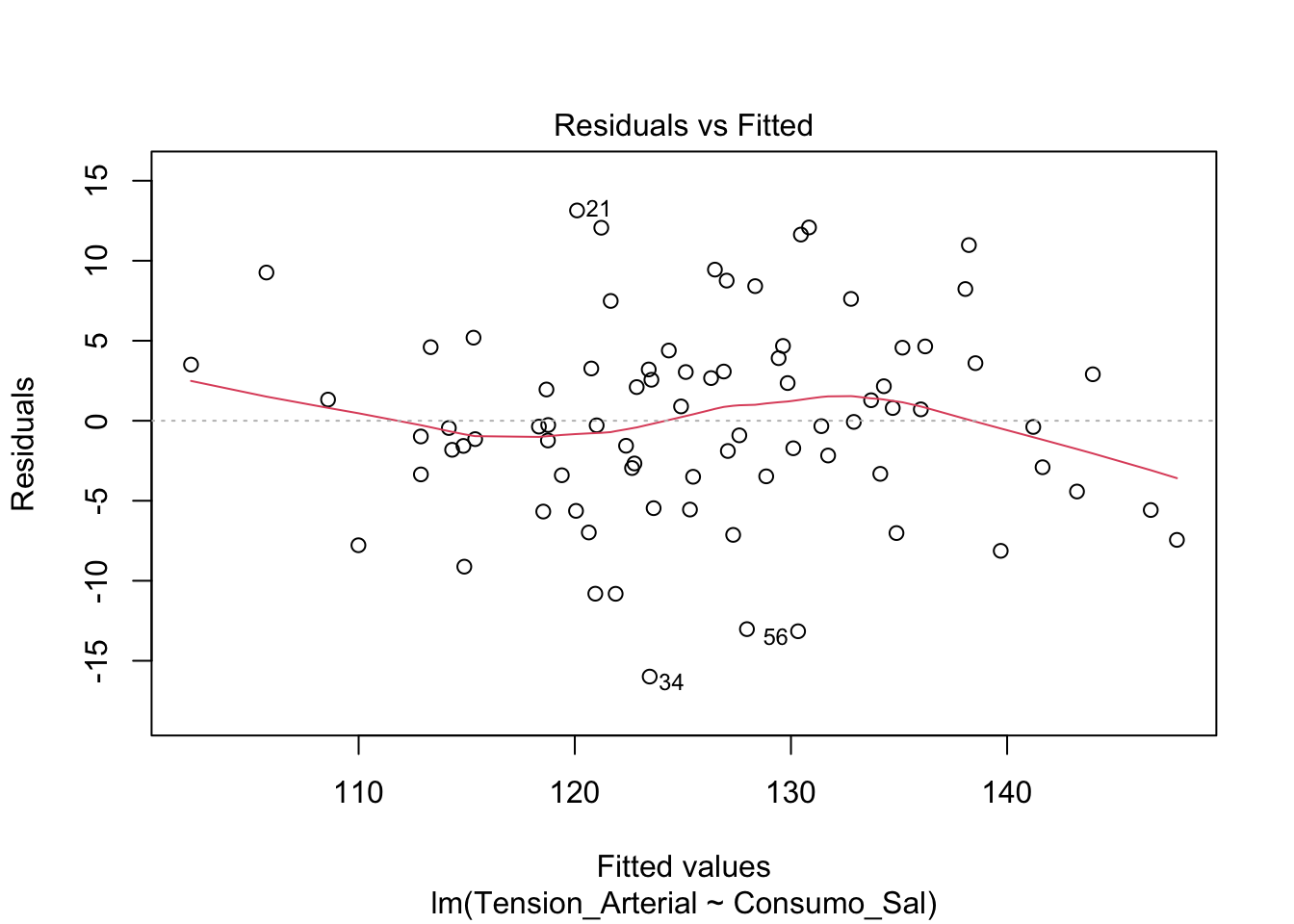
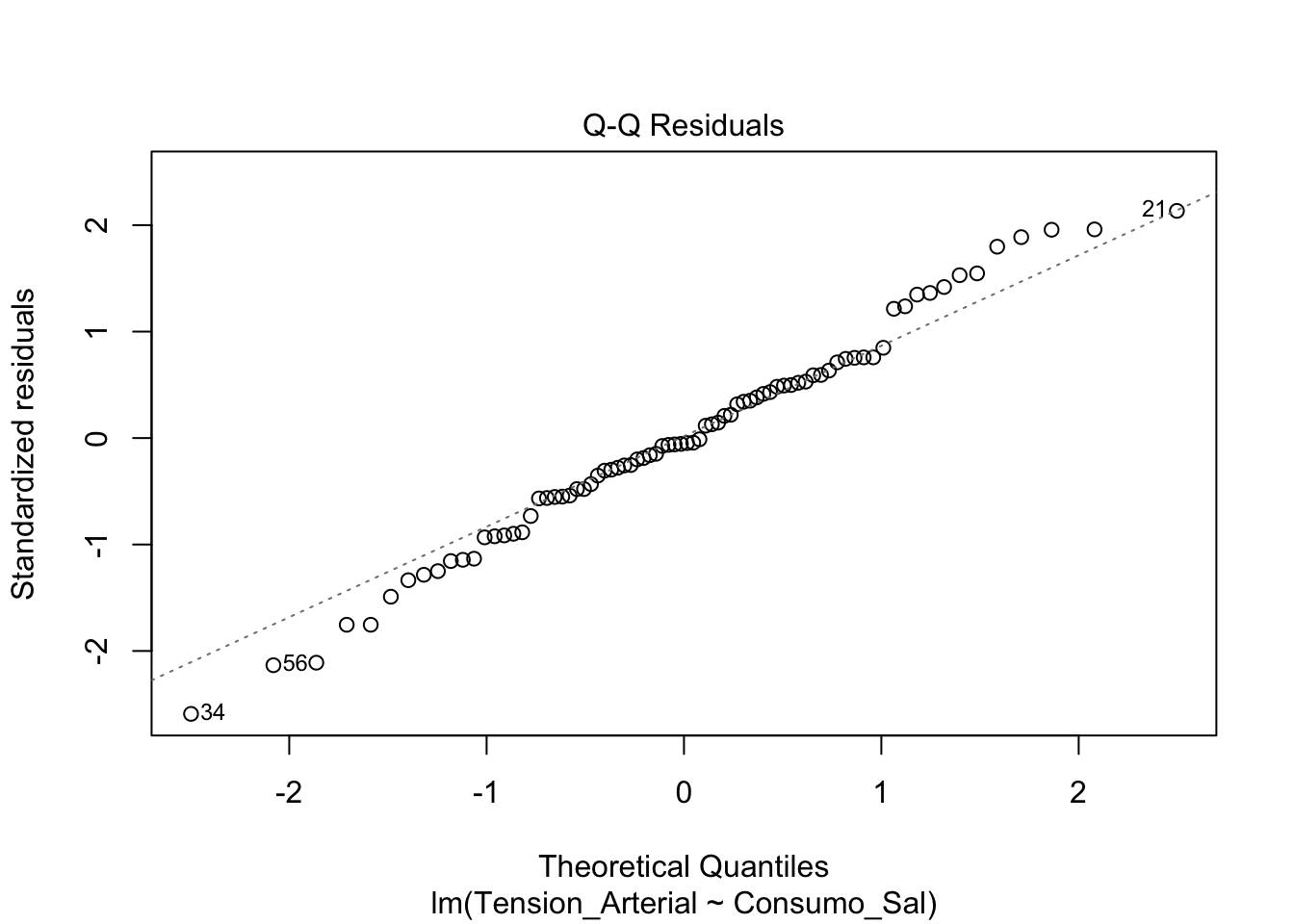
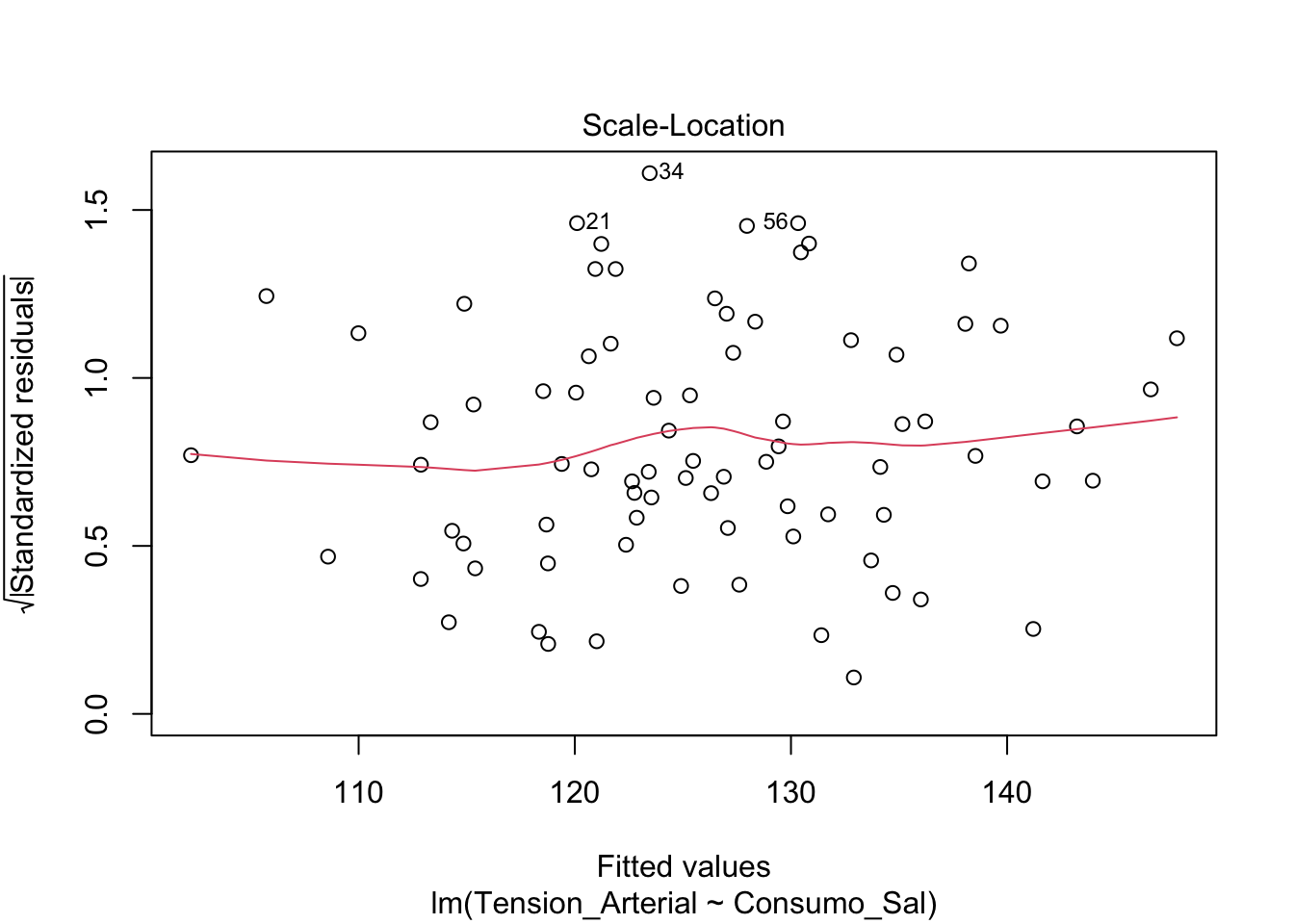
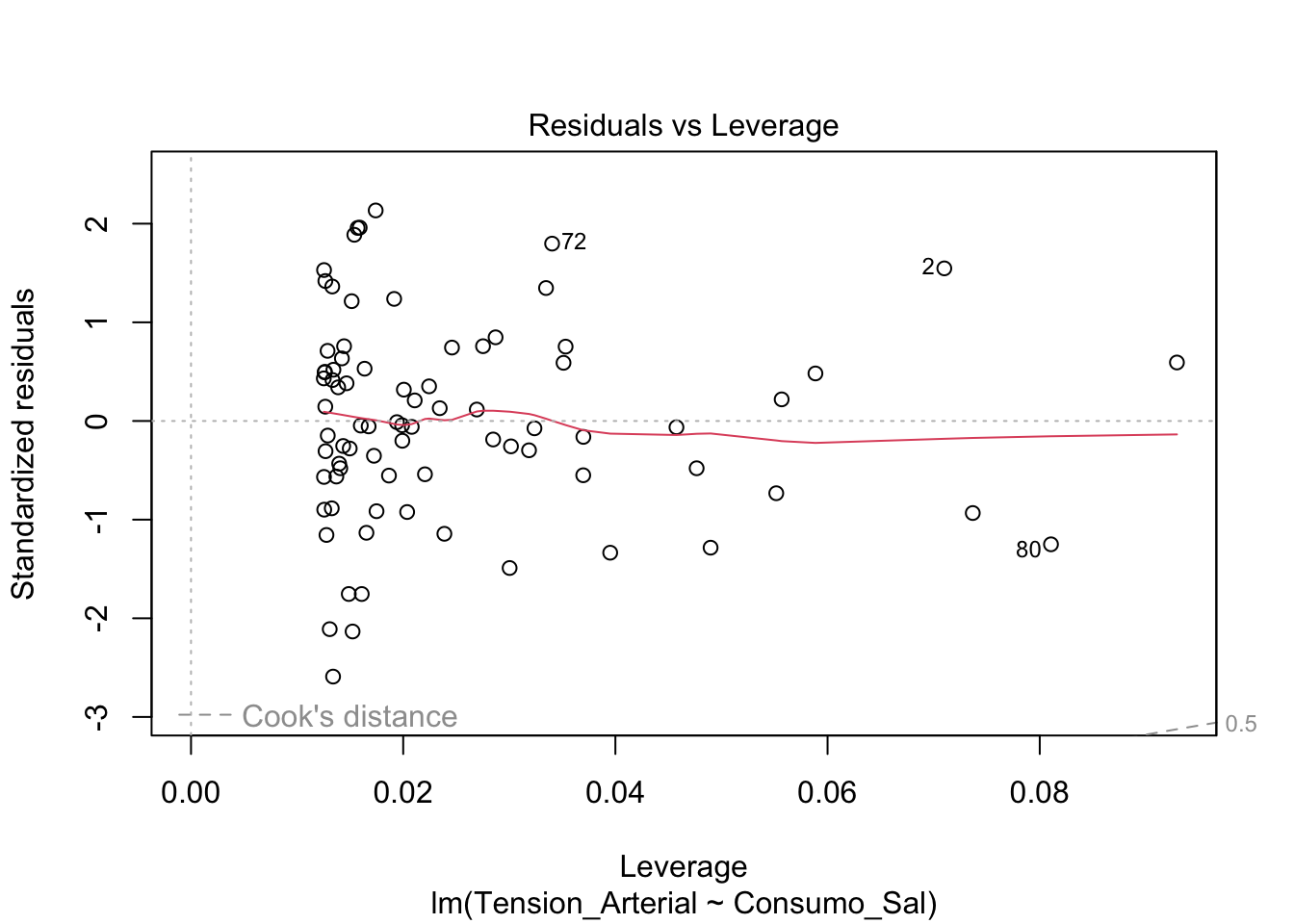
Uitlizando el perfromance también podemos evaluar los supuestos del modelo.
performance::check_model(modelo)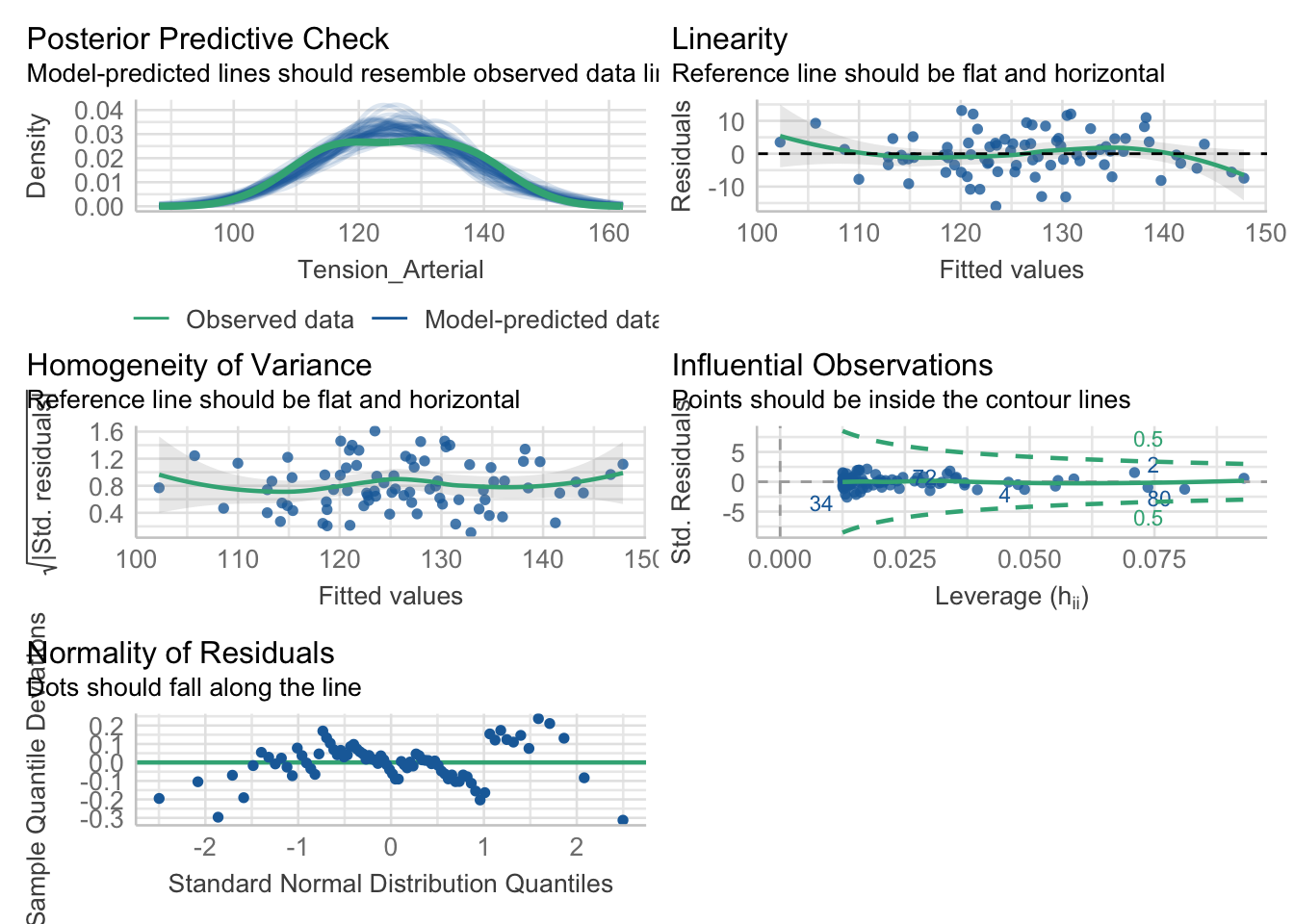
Resumen de los supuestos
Se hace una recopilación de los supuestos evaluados en el ejemplo de la regresión.
- Linealidad: Debe de evidenciarse una relación lineal entre las variables antes realizar un modelo de regresión lineal
- Utilice gráficos de dispersión
- Coeficiente de correlación de Pearson
- Independencia de los datos. Este concepto no se había explicado antes, sin embargo, es fácil de comprender. En una regresión las observaciones individuales no deben estar relacionadas o influenciadas entre sí.
- Cuando se recolectan los datos debe de asegurarse la no relación de la variables, por ejemplo, no se debe de incluir mediciones repetidas del mismo sujeto • En los gráficos de residuales también se hace evidente esta relación
- Homoscedasticidad: los residuos tienen varianza constante a lo largo de los valores predichos.
- Utilice gráficos de diagnóstico para comprobar la homoscedasticidad.
- Normalidad de los Residuos: los residuos del modelo deben de estar distribuidos normalmente.
- Histogramas de los residuos.
- Gráficos Q-Q de los residuos.
- Pruebas de normalidad
Predicciones
Con una regresión podemos dado un dato de la variable independiente predecir el valor de la variable dependiente. Por ejemplo
predict(modelo, newdata = data.frame(Consumo_Sal = 10)) 1
75.0428 Un mal ejemplo
Se desea comprobar la dependencia de la edad sobre la adiponectina. Construya un modelo de regresión lineal utilizando la base de datos SLE Dataset 3
library(readr)
SLE_dataset3 <- read_csv("Bases/SLE dataset3.csv")- Graficar
plot(SLE_dataset3$Adiponectin~ SLE_dataset3$Age, main="Concentraciones de adiponectina por edad")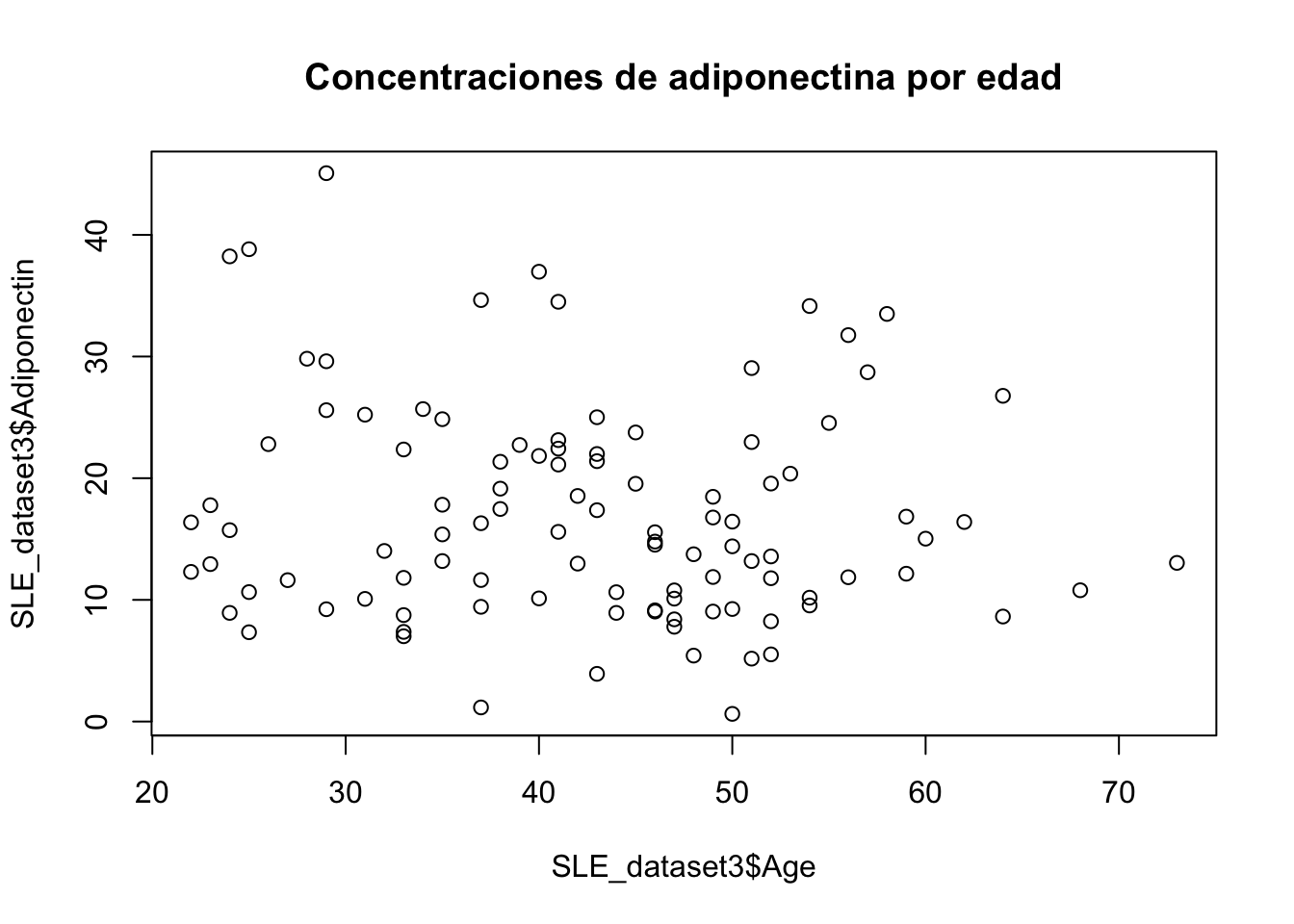
cor.test(SLE_dataset3$Adiponectin,SLE_dataset3$Age)
Pearson's product-moment correlation
data: SLE_dataset3$Adiponectin and SLE_dataset3$Age
t = -1.2614, df = 101, p-value = 0.2101
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.31057423 0.07069386
sample estimates:
cor
-0.1245346 - Construir objeto
mod2 <- lm(SLE_dataset3$Adiponectin~ SLE_dataset3$Age)- Resultados del modelo
summary(mod2)
Call:
lm(formula = SLE_dataset3$Adiponectin ~ SLE_dataset3$Age)
Residuals:
Min 1Q Median 3Q Max
-16.372 -6.577 -1.860 4.818 26.754
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 21.15562 3.41905 6.188 1.32e-08 ***
SLE_dataset3$Age -0.09792 0.07763 -1.261 0.21
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 8.832 on 101 degrees of freedom
Multiple R-squared: 0.01551, Adjusted R-squared: 0.005761
F-statistic: 1.591 on 1 and 101 DF, p-value: 0.2101- Interpretación
- La adiponectina es independiente de la edad. El ANOVA nos dice que el modelo no es adecuado ya que la variación de los residuales es muy alta
- El coeficiente \(\beta_1\) no es siginifativo por lo tanto no podemos asegurar que sea distinto de cero
- Validez del modelo
plot(mod2)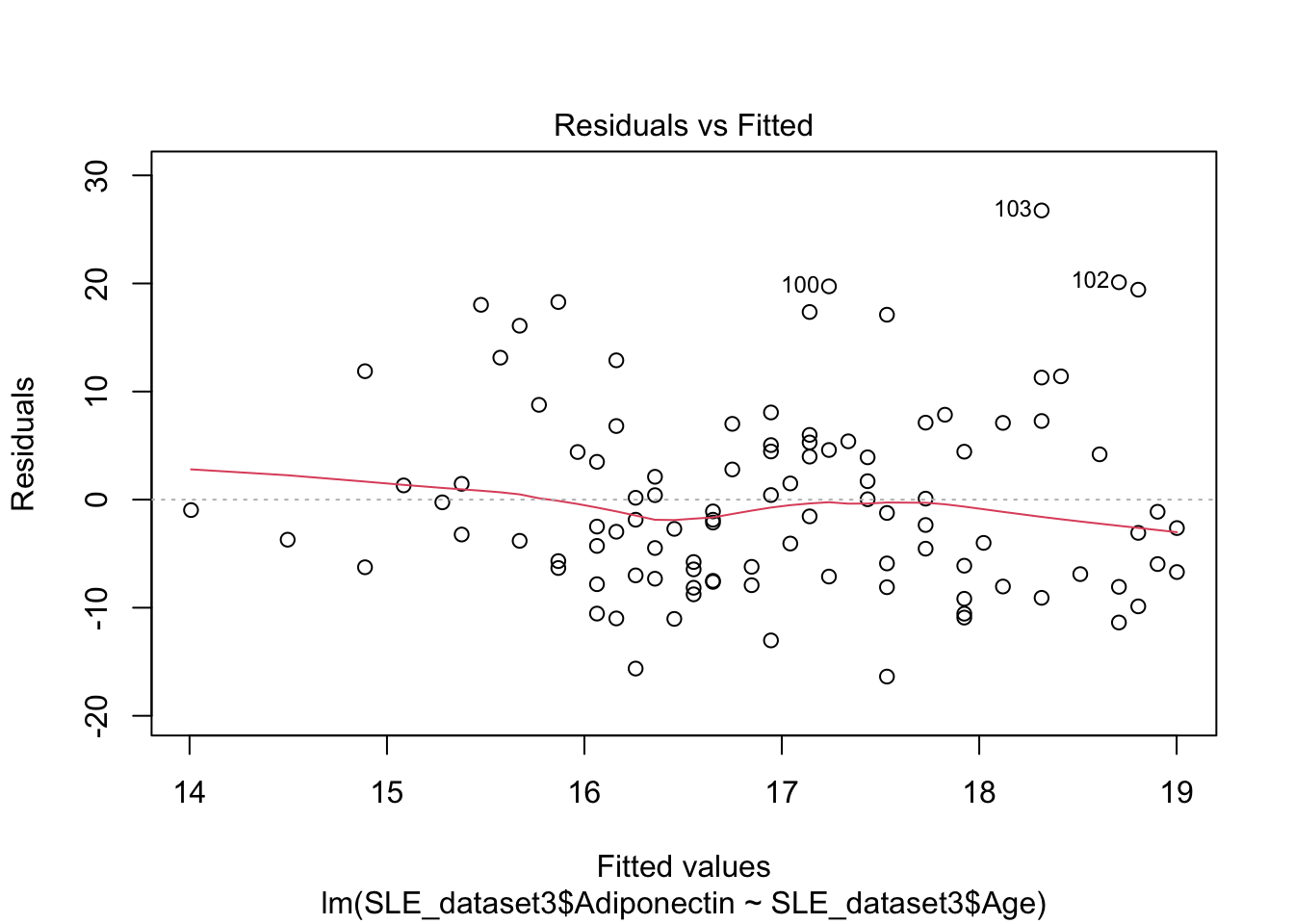
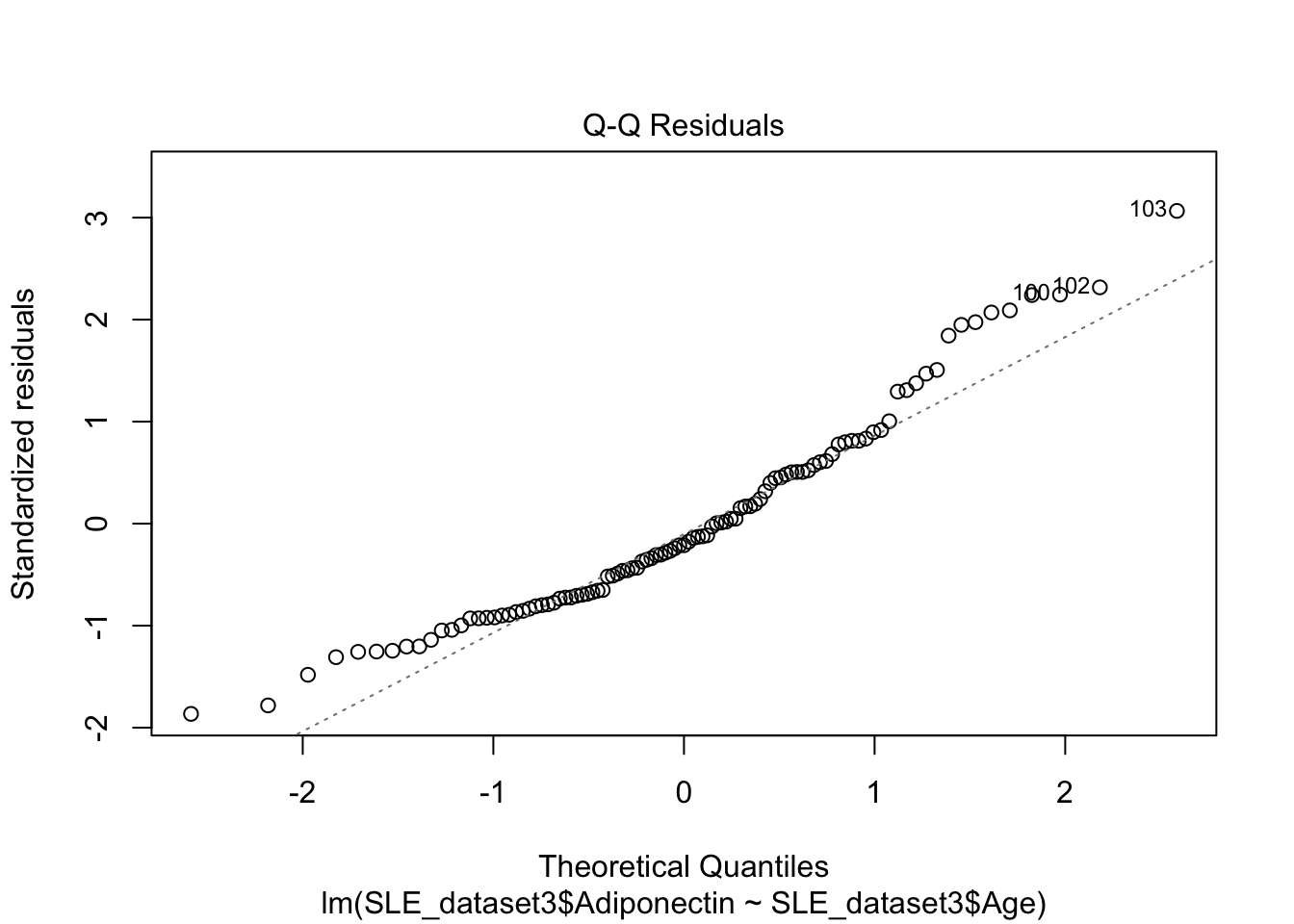
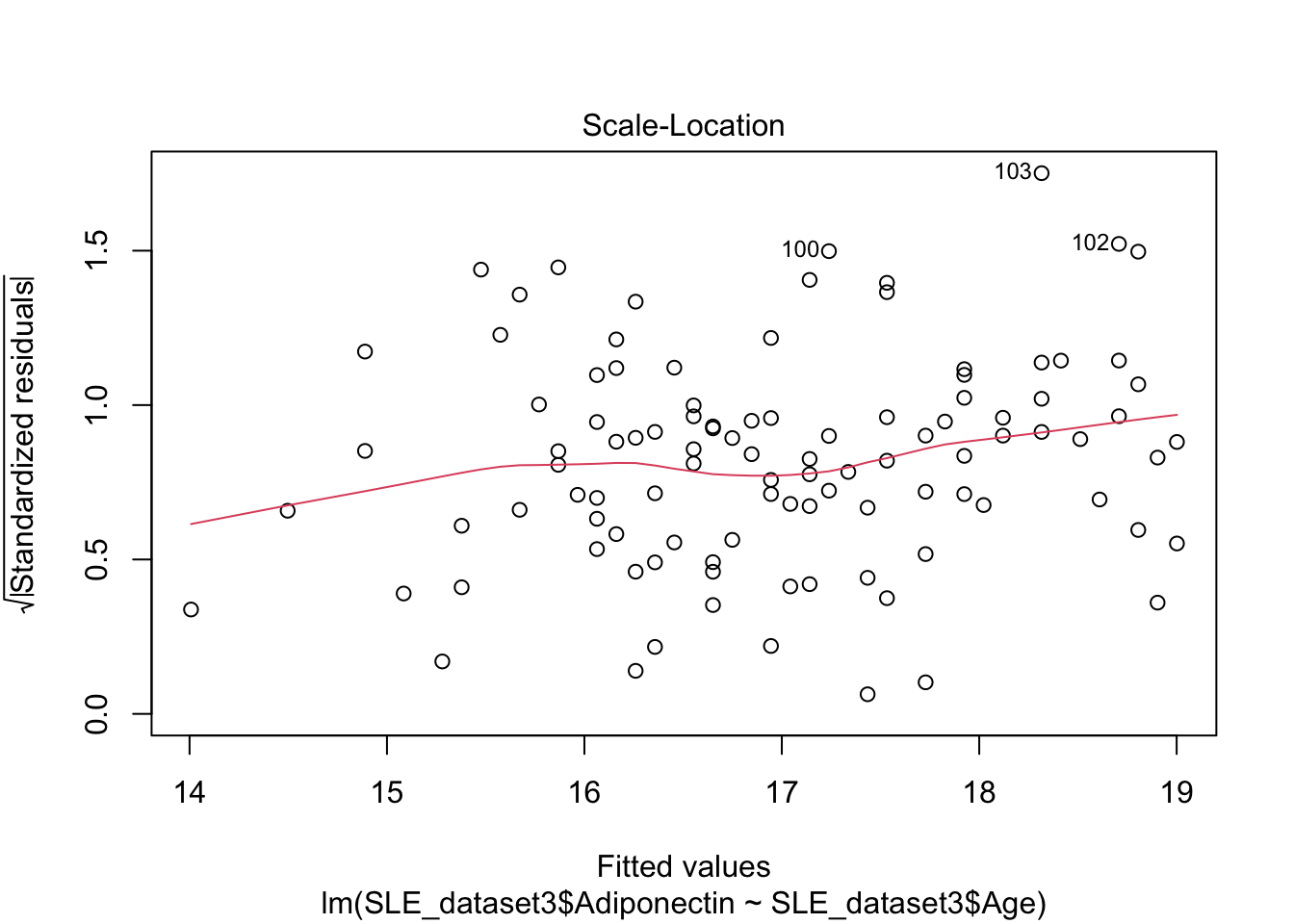
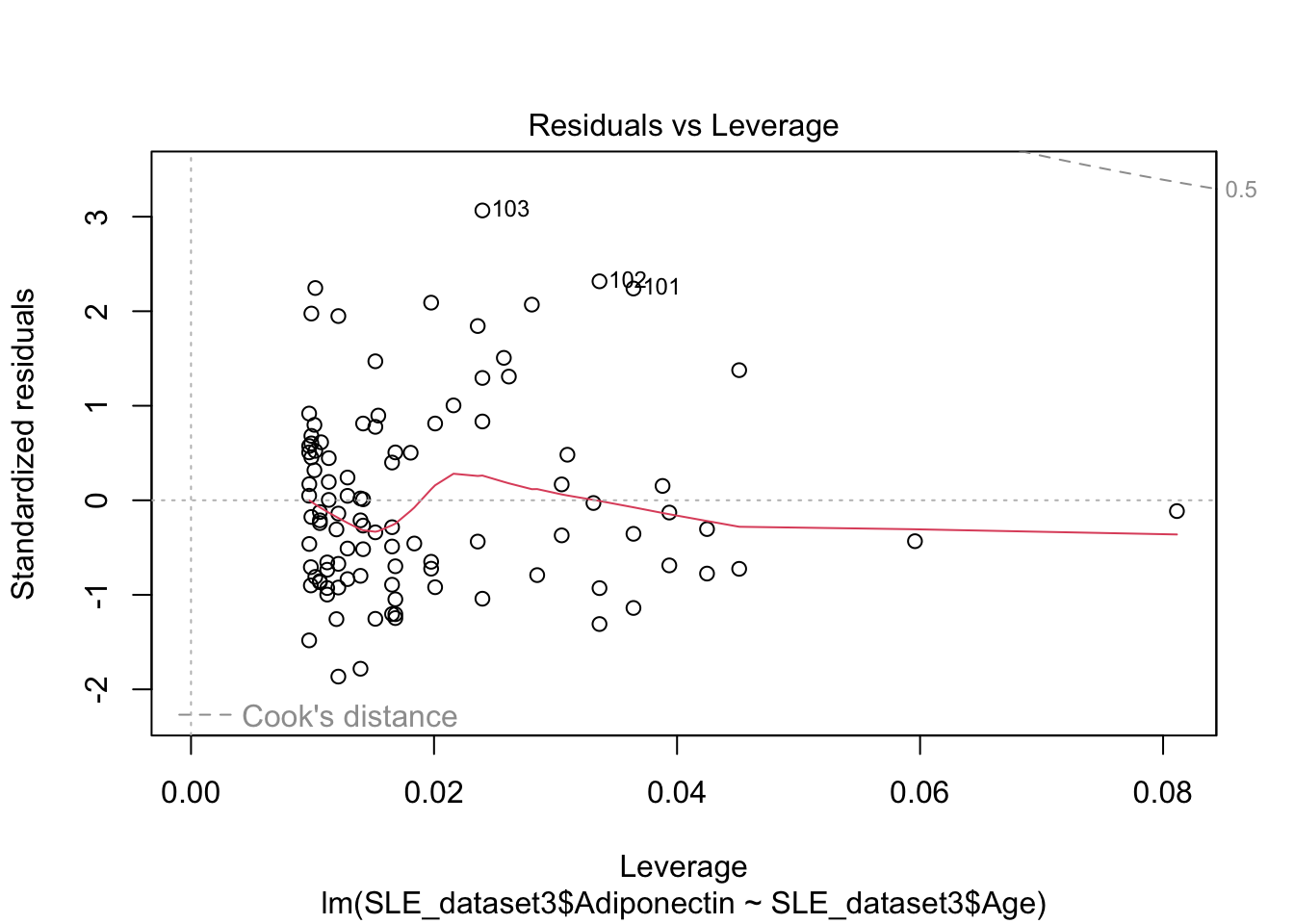
El gráfico Q-Q muestra que los residuos no se apegan a una distribución normal. Por tanto nuestro modelo no es valido.